
新冠病毒(COVID-19)自2019年底首次爆发以来,已造成全球大流行,全球范围内的抗疫工作依赖于疫苗接种、病毒检测及药物治疗等手段。虽然目前已有几种疫苗和抗病毒药物上市,但新冠病毒变异株的出现以及对抗药物的耐药性使得新的药物研发仍然迫在眉睫。了解突变对病毒蛋白三维结构以及分子识别和相互作用的影响,对研究病毒生命周期的分子机制具有重要意义,也可能为药物和疫苗设计提供线索。
为了快速筛选可能的抗病毒药物,研究人员依赖于计算机辅助药物设计(CADD)技术,尤其是分子对接(molecular docking)方法。分子对接可以模拟小分子药物与病毒靶标之间的结合模式,预测其结合亲和力,从而筛选出潜在的有效药物。然而,鉴于新型冠状病毒的快速变异特性,传统的药物筛选方法因耗时冗长且需大量实验验证,已不再适用。在这里,我们将介绍一个用于预测COVID-19靶点与潜在配体结合模式的工具——nCoVDock2,它能够为研究人员提供快速、精确的分子对接计算,从而加速抗新冠病毒药物的发现。
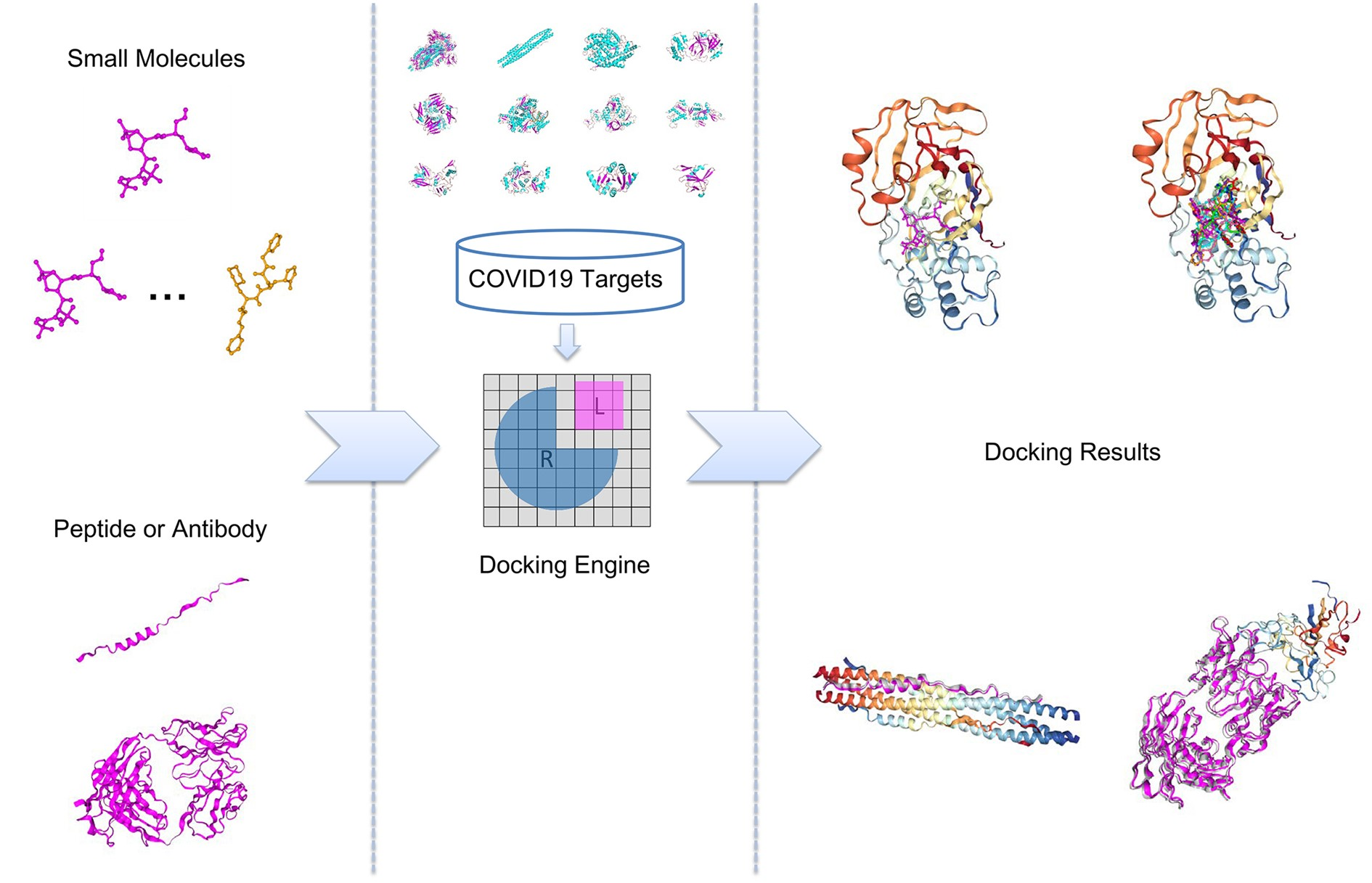
nCoVDock2(https://ncovdock2.schanglab.org.cn)不仅可以预测COVID-19靶点与小分子配体之间的结合模式,同时还可以将多肽和抗体作为配体进行对接研究。其内置靶标库包括了多种SARS-CoV-2病毒靶标(PLpro、Mpro、S蛋白等各种野生型与突变型结构)。Autodock Vina作为对接引擎,用于小分子对接。除了Autodock Vina评分,该工具还提供了一个基于机器学习的打分函数RF-Score作为补充来评估小分子的结合亲和力。在肽或抗体对接时,CoDockPP(在CASP-CAPRI国际生物大分子结构预测比赛中表现优异)作为对接引擎,还增加了一个基于深度神经网络的打分函数来进行来进行更为精确和高效的亲和力评估。
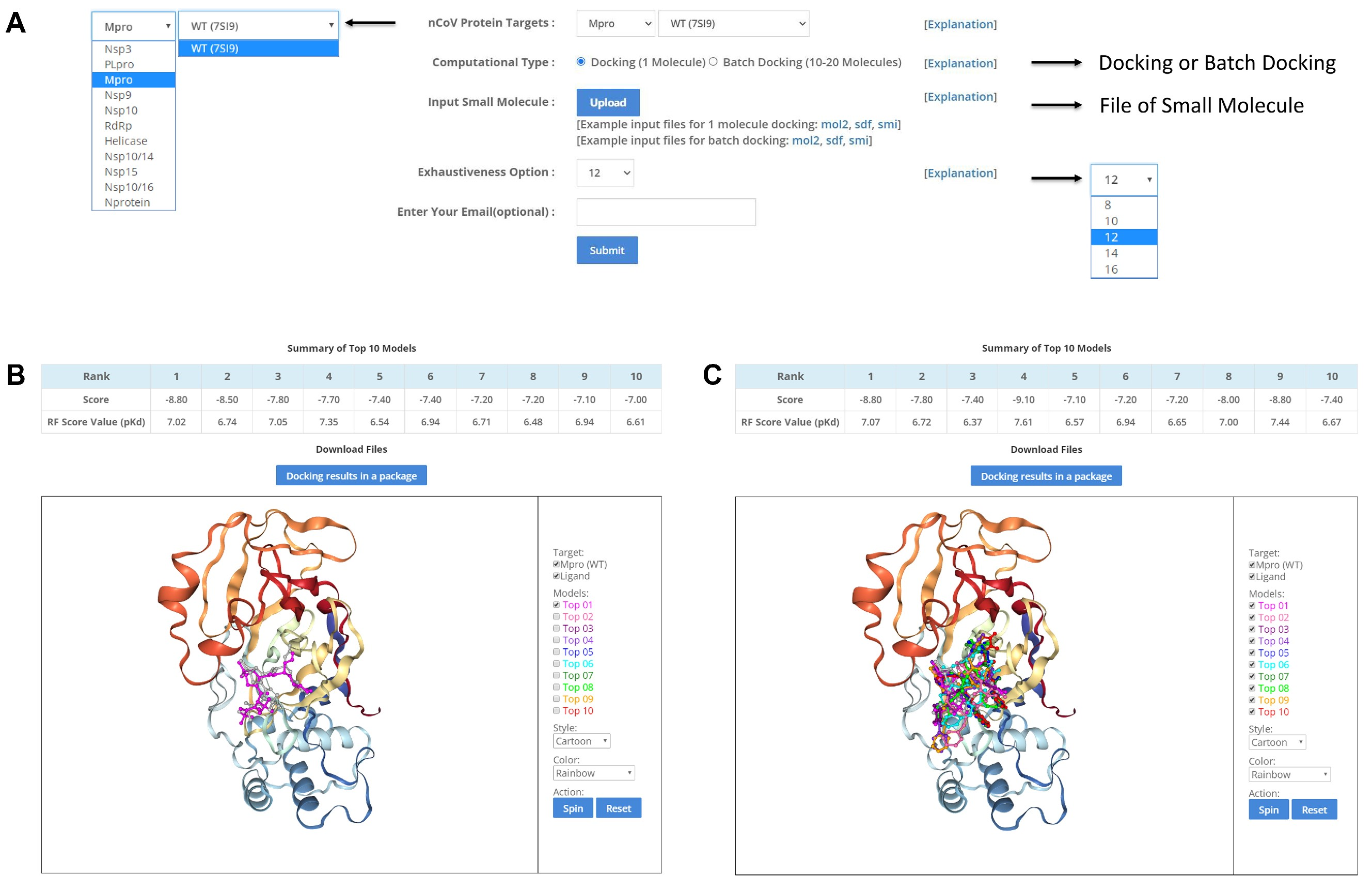
如图A所示,用户首先需要从窗口选择感兴趣的COVID-19靶点及其野生型或突变体。小分子对接有两个计算模块。如果选择“Docking”,则只需上传一个小分子,结果页面将显示该分子对接打分排名前10的结合模式。如果选择“Batch Docking”,则需要上传10-20个分子,结果页面将显示打分排名前10位的分子。这些小分子可以以smi、mol2或sdf格式上传。Exhaustiveness是对接搜索的一个参数,该值越大计算时间越长。用户可提供一个电子邮件地址来接收作业提交和作业状态的消息。作业完成后,会向用户的邮箱发送通知邮件。
图B显示的是单分子对接结果,表格提供了该分子对接打分排序前10的结果,包括Vina打分结果(Score,值越小说明结合亲和力越强)和RF Score打分结果(值越大说明结合亲和力越强)。结合构象可以通过页面上的NGL分子视图来观察,也可以对蛋白显示Style以及Color进行选择。图C显示的是多分子对接结果,表格提供了打分排名靠前的10种小分子,分子视图中10种小分子的预测结合姿势用不同的颜色表示。

与小分子对接类似,用户需要先选择COVID-19蛋白靶点(图A),然后以pdb格式上传多肽或抗体蛋白。该工具提供全局对接和位点特异性对接两种方式,用来预测靶蛋白与配体蛋白的结合模式。在文本框中,用户可以在靶蛋白界面和配体蛋白界面上分别输入一个约束残基。如果只定义配体或靶蛋白上的一个残基,则保留复合物界面上该特定残基的构象。当用户同时定义受体上的一个残基和配体上的另一个残基时,则需要选择约束类型:模糊约束(Ambiguous)还是多重约束(Multiple)。当选择模糊约束时,在界面上保留至少一个选定的残基构象。当选择多个约束条件时,在界面上的两个残基都保留构象。
图B和图C分别显示了多肽和抗体的对接结果,表格提供了该分子对接打分排序前10的结果,包括CoDockPP打分结果(Score,值越小说明结合亲和力越强)和基于深度神经网络的打分结果(AI Score,值越小说明结合亲和力越强)。同样,蛋白-多肽、蛋白-抗体的结合构象可以通过页面上的NGL分子视图来观察。
本文构建的nCoVDock2靶点筛选系统,可以用于预测野生型和突变型SARS-CoV-2治疗靶点及其潜在配体(小分子、多肽、抗体)之间的结合模式。该服务器为预测结果提供了友好的界面和结合模式可视化,使其成为COVID-19药物发现的有用工具。
Liu K, Lu X, Shi H, et al. nCoVDock2: a docking server to predict the binding modes between COVID-19 targets and its potential ligands[J]. Nucleic Acids Research, 2023, 51(W1): W365-W371.