
MicroRNAs (miRNAs)通过miRNA沉默过程调节植物和动物中数千种基因的表达,miRNA沉默过程参与多种重要的生理过程,包括上皮再生、心脏功能、排卵和癌症发展。Argonaute 2(Ago2)蛋白是miRNAs引导的基因抑制和沉默过程的主要载体,miRNA通过与Ago2形成RNA诱导沉默复合物(RNA- induced silencing complexes, RISCs),可作为识别互补靶mRNA的向导,并通过翻译抑制和降解引起特异性基因沉默。虽然人类Ago2(hAgo2)的晶体结构近年来已被揭示,但其与RNA识别的动态结构特征信息以及相应的蛋白质结构变化信息仍然缺乏。
本文采用分子动力学(MD)模拟的方法研究了hAgo2-RNA(有RNA结合)体系和hAgo2-free(无RNA结合)体系的动力学特性,为进一步了解Ago2-RNA识别的分子机制提供依据。
在这项研究中,研究人员构建了Ago2两种状态的模型:一个是与RNA结合的复合物体系(hAgo2-RNA),另一个是没有RNA结合的游离Ago2体系(hAgo2-free)。然后,进行了长时间的分子动力学模拟,并对模拟结果进行了多种分析,以探讨Ago2在这两种状态下的结构稳定性、灵活性、相互作用等动力学特征。通过比较这两个体系的动态特征,揭示了Ago2在RNA结合过程中的结构变化和功能性运动。
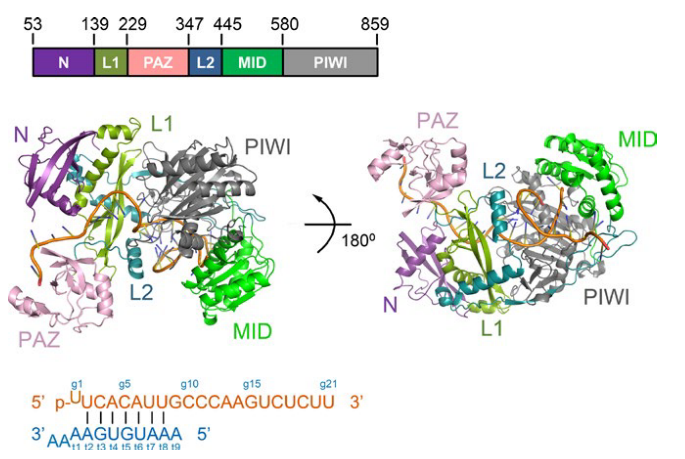
hAgo2和RNA复合物的晶体结构来源于PDB数据库 (PDB ID: 4W5Q)。其中hAgo2由四个球状结构域(N、PAZ、MID、PIWI)和两个链接结构域(L1和L2)组成,RNA包括guide RNA(g1-g21)和target-RNA(t1-t9)。由于蛋白质数据库中没有完整的游离hAgo2结构,因此从复合物结构中去除RNA结构以获得hAgo2-free体系。随后对构建的两个体系分别进行100 ns的分子动力学模拟。
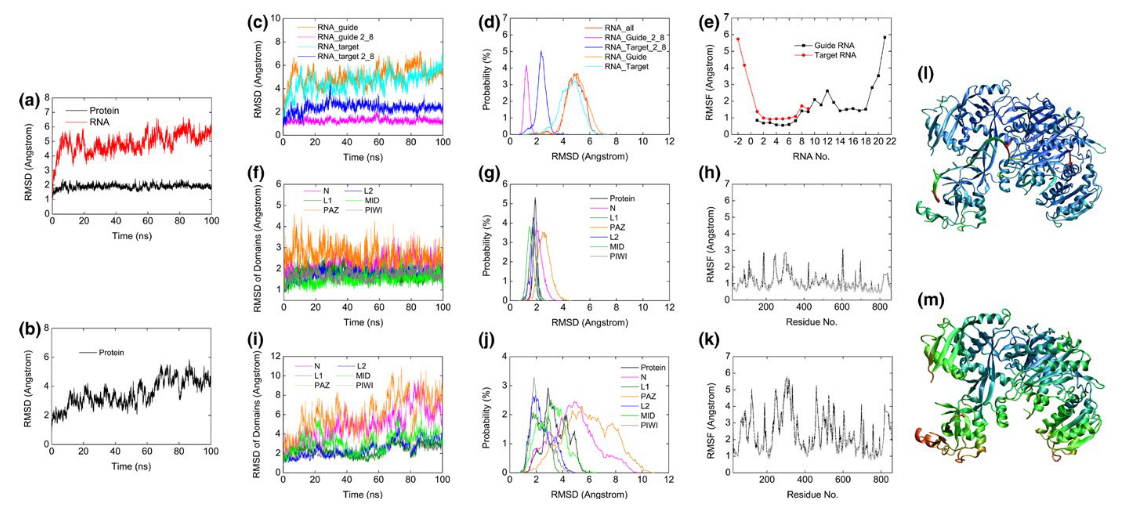
RMSD是衡量分子在模拟过程中与初始结构之间偏差的标准指标。通过计算RNA结合和未结合Ago2的RMSD,研究者可以评估其结构的稳定性。Ago2-RNA体系中,蛋白的RMSD值在模拟过程中没有大幅波动,意味着蛋白的结构稳定保持在一个合理的范围内。在没有RNA结合的体系中,Ago2显示出较大的RMSD波动,表明Ago2结构较为灵活,RNA去除后蛋白发生了强烈的结构重排。
RMSF反映了蛋白质各个氨基酸残基的波动程度,可以揭示蛋白质结构中的柔性区域。没有RNA结合时,Ago2的结构显示出较高的RMSF(图k、m),特别是在一些连接区域和表面区域,表明这些部分更加灵活,结构稳定性较差。RNA结合后,Ago2的某些区域(如PIWI结构域)表现出较低的RMSF(图h、l),表明这些区域的运动较为受限,可能因为RNA的结合导致这些区域更加稳定。
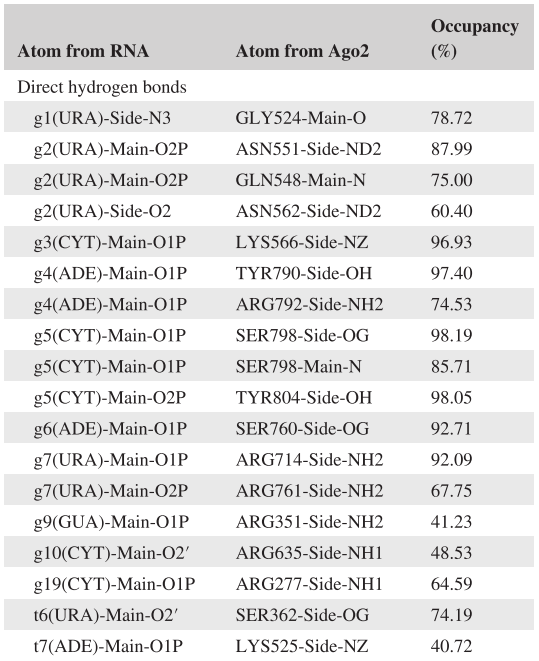
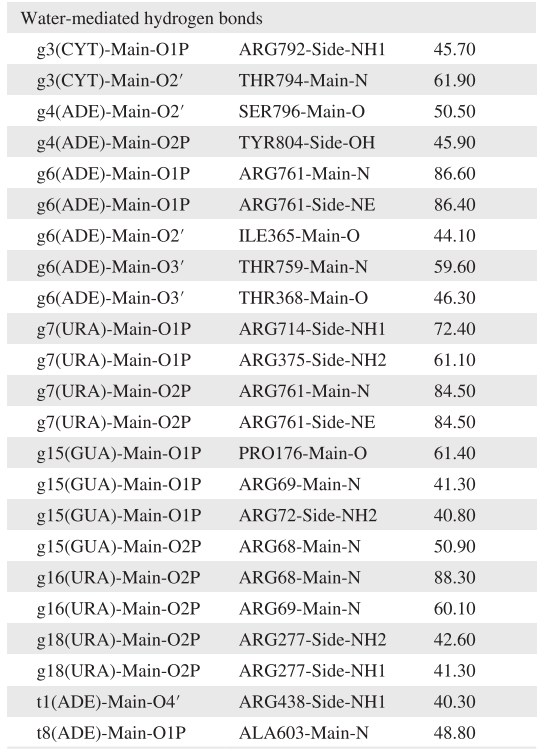
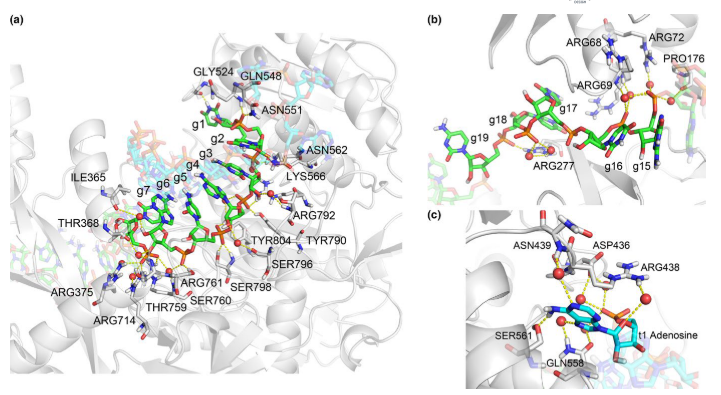
氢键分析有助于了解分子之间的相互作用以及蛋白质内部的稳定性。在hAgo2-RNA体系中,观察到了18个强的直接氢键和23个水介导的氢键,主要分布在g2–g7段和RNA骨架与蛋白之间。这表明蛋白与RNA的相互作用主要由RNA骨架原子介导,与Ago2与miRNA的非特异性识别相一致。
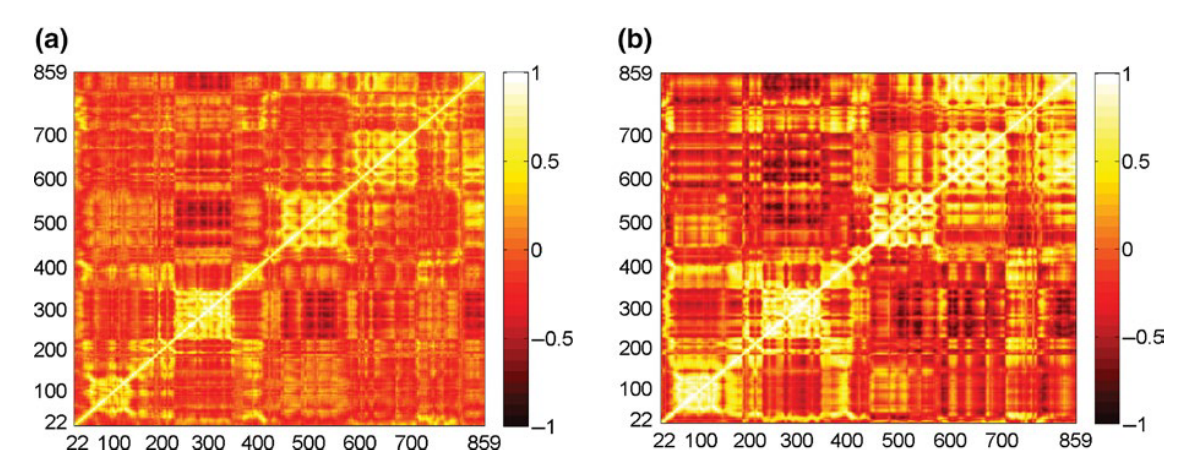
DCCM(动力学交叉相关图)分析能够揭示蛋白质不同区域之间的协同运动或反向运动。为了研究hAgo2的内在结构特性,利用DCCM揭示了hAgo2两种状态下的相关运动(a:hAgo2-RNA、b:hAgo2-free)。颜色条表示从白色到红色再到黑色,相关系数从1(极正相关)到0(不相关)到−1(极负相关)。两幅图中的白色对角线表明,每个残基的运动与自身绝对正相关。从图中可以看出四个浅色方块位于对角线上,它们对应于hAgo2中的四个全局结构域:N(残基22-138)、PAZ(残基229-346)、MID(残基445-579)和PIWI(残基580-859)。在两个体系中,残基220-330和残基450-580之间存在很强的负相关,它们分别由PAZ和MID结构域组成。这表明PAZ和MID结构域在两个体系中进行相反方向的运动。在hAgo2-free体系中,正相关和负相关显著增强,表明hAgo2- free体系的构象变化更大。此外,N和PAZ域(残基22-450)与自身呈正相关,而与MID和PIWI域(残基451-859)呈负相关。这表明蛋白可以被分为两部分,这两部分在有或没有RNA结合时总是向相反方向移动。

PCA分析用于提取系统的基本运动模式。结果显示Ago2蛋白在有或无RNA结合状态下明显表现出不同的运动模式(a:hAgo2-RNA、b:hAgo2-free)。在hAgo2-RNA系统中,除了PAZ和PIWI/MID之间的轻微开放运动外,短得多的锥体长度意味着RNA结合的整个结构的稳定性。在hAgo2-free体系中,随着锥体长度的增加,检测到的构象变化更大,运动方向似乎与RNA结合体系相反,尤其是PAZ结构域。
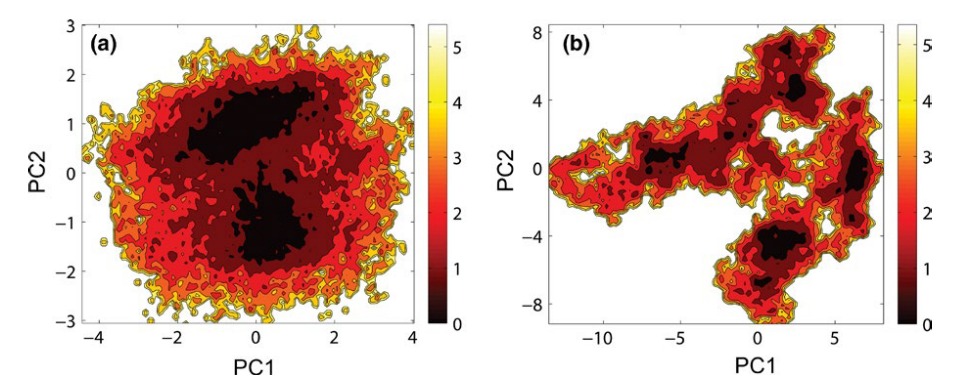
自由能景观(FEL)分析有助于揭示复合物在不同构象下的稳定性,颜色越深表示能量越低。从图中可以看出(a:hAgo2-RNA、b:hAgo2-free),hAgo2-RNA体系主要存在两种低能构象状态,hAgo2-free低能状态更多,构象变化较为丰富。
本研究通过分子动力学模拟方法,详细研究了hAgo2与RNA的结合过程,从RMSD、RMSF、氢键分析、DCCM、PCA、FEL等多个方面出发,深入分析了Ago2-RNA识别的分子机制。基于研究结果推测PAZ结构域动态构象改变可能是hAgo2介导的miRNA诱导基因调控的关键因素。这些发现为理解Ago2在RNA干扰中的作用提供了深入的理论基础,并有助于未来在miRNA研究和基因表达调控方面的进一步探索。该研究成果已发表于学术期刊《Chemical biology & drug design》。
Kong R, Xu L, Piao L, et al. Exploring the RNA‐bound and RNA‐free human Argonaute‐2 by molecular dynamics simulation method[J]. Chemical biology & drug design, 2017, 90(5): 753-763.