
Shank3(SH3 and multiple ankyrin repeat domains protein 3)是一种调控突触信号传导的蛋白,与22q13缺失综合征和自闭症谱系障碍(ASD)的发生密切相关。SAPAP(synapse-associated protein 90/postsynaptic density-95-associated protein)是与突触功能相关的蛋白,在神经系统中扮演重要角色。SAPAP通过其扩展的PDZ结合模体(E-PBM)与Shank3的N-PDZ结构域结合,在突触可塑性和神经信号传递中起着关键作用。生物化学和结构实验为Shank3和SAPAP结合提供了基础知识基础。然而,Shank3和SAPAP(野生型和突变型)之间相互作用的分子机制尚未完全阐明。
本文采用了分子动力学(MD)模拟和MM/GBSA(Molecular Mechanics/Generalized Born Surface Area)结合自由能计算的方法来研究SAPAP与Shank3之间结合的分子机制,特别是野生型和突变型E-PBM与Shank3 N-PDZ之间的相互作用差异。
在这项研究中,研究人员构建了SAPAP的野生型E-PBM和两种突变型E-PBM(M4P和M5P)与Shank3 N-PDZ的复合物模型。M4P是将E-PBM的C端部分(974QTRL977)突变为四个脯氨酸,而M5P是将N端部分(966IEIYI970)突变为五个脯氨酸。通过MD模拟来研究Shank3与野生型SAPAP E-PBM以及两种突变肽之间结合的分子机制。结合动态互相关图(DCCM)分析、主成分分析(PCA)和自由能景观(FEL)方法探讨了突变引起的构象变化等动力学特性。使用分子力学/广义玻恩表面积(MM/GBSA)方法来评估结合自由能。该研究揭示了Shank3与SAPAP特异性结合的分子机制,并为设计肽抑制剂干扰它们之间的相互作用提供了线索。
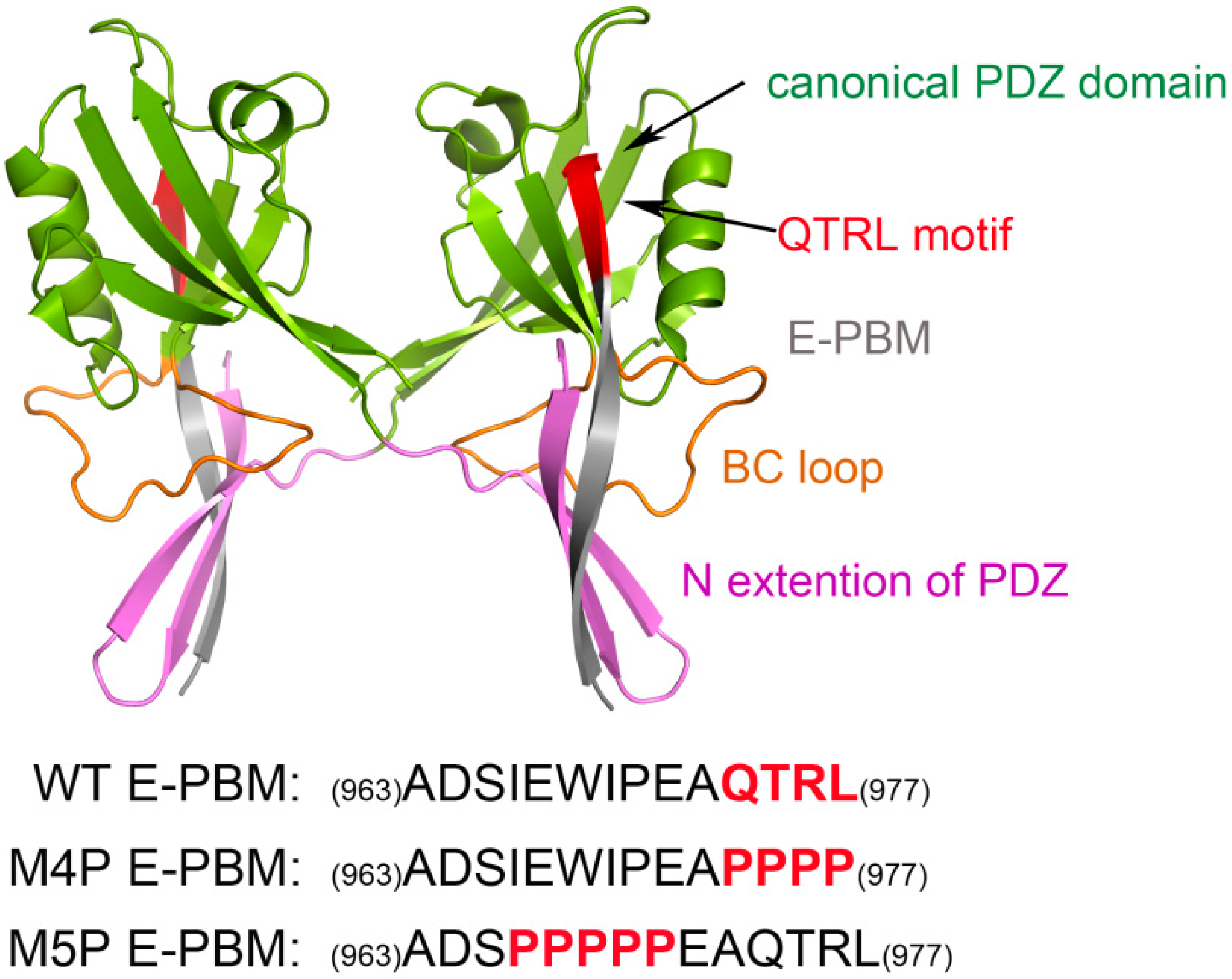
Shank3 N-PDZ和SAPAP E-PBM的结构来源于PDB数据库 (PDB ID: 5IZU)。为了与实验数据进行比较,将QTRL(974-977)和IEIWI(966-970)的序列分别替换为脯氨酸,构建了两个突变体系。随后对构建的三个体系分别进行100 ns的分子动力学模拟。
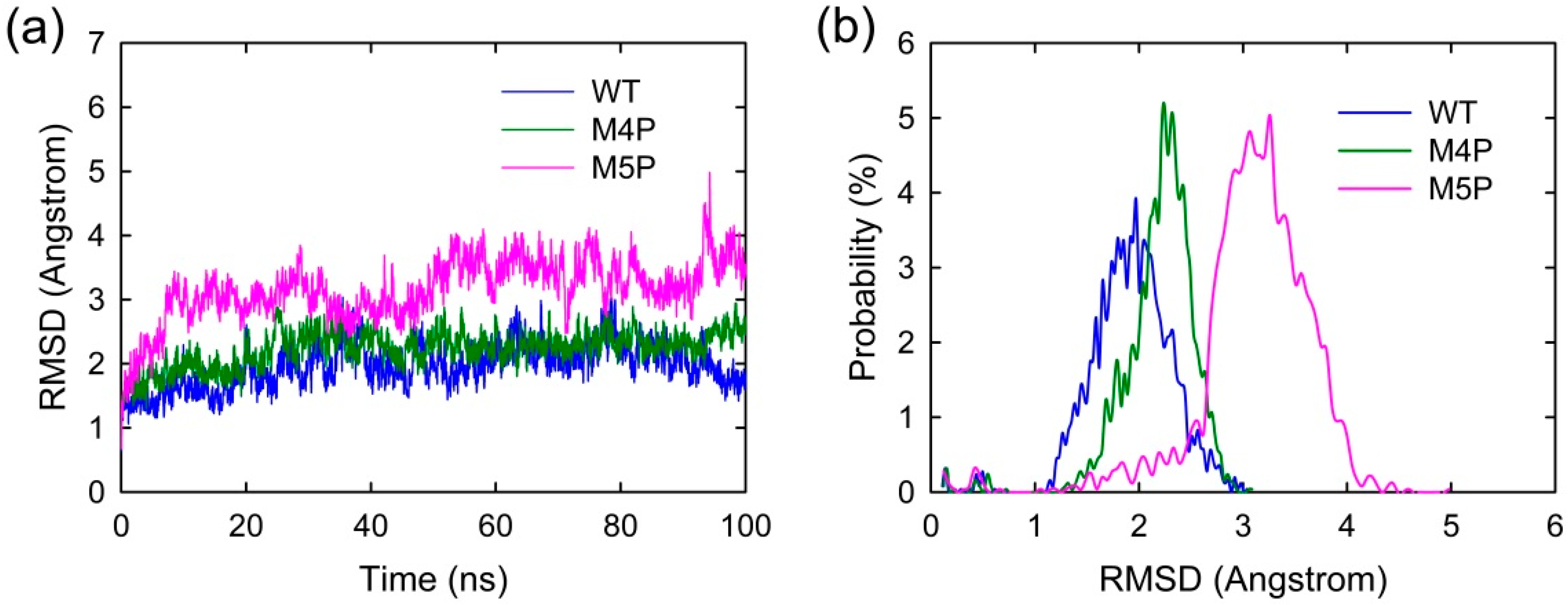
RMSD用于评估模拟过程中蛋白质结构的稳定性。在所有系统中,RMSD值在20 ns模拟后收敛,表明系统达到了稳定状态。然而,在突变体系中(特别是M5P系统),RMSD值持续增加,这意味着突变可能导致结构扰动和构象重排,影响了复合物的稳定性,降低了SAPAP与Shank3的相互作用强度。
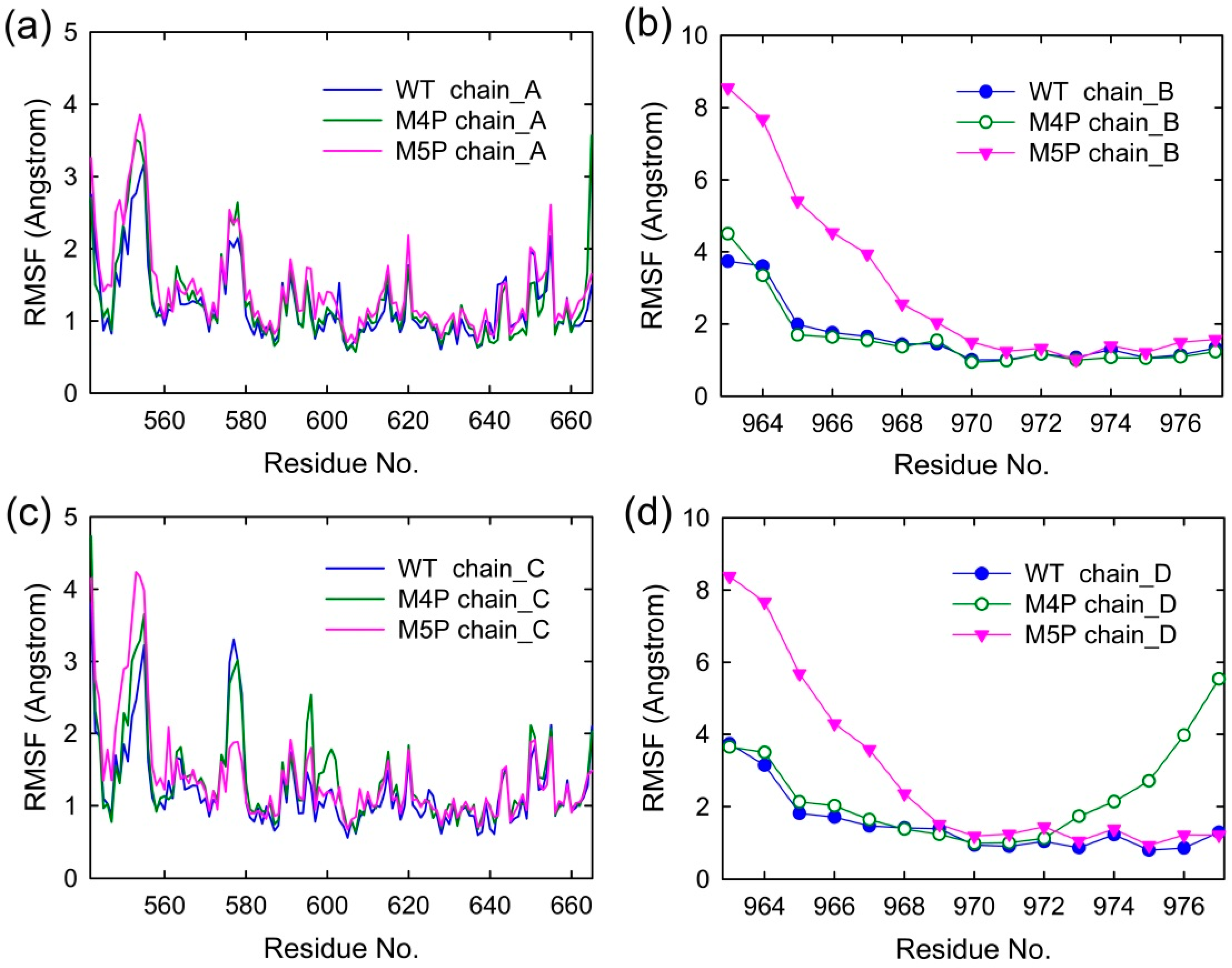
RMSF反映了原子位移的幅度,即蛋白质的柔韧性。在野生型系统中,除了几个特定区域外,canonical PDZ domain(564-665)的RMSF值较低,表明其结构相对稳定。相比之下,M4P系统中E-PBM的C端残基(973-977)和M5P系统中E-PBM的N端残基(963-968)及βN发夹结构域(542-563)的RMSF值显著增加,表明这些区域的柔韧性增加,可能导致结合亲和力的降低。
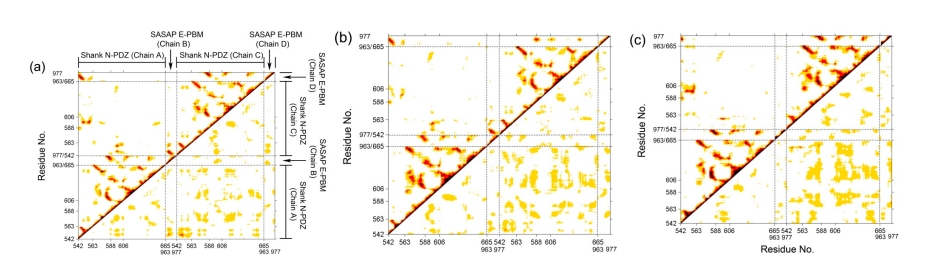
DCCM分析用于研究Shank3和SAPAP之间的相关运动。结果显示,在野生型系统中,E-PBM与canonical PDZ domain和βN发夹结构域之间存在强烈的正相关运动。然而,在M4P系统中,E-PBM与canonical PDZ domain之间的正相关运动减弱,而在M5P系统中,这种正相关运动几乎消失。这表明突变影响了E-PBM与Shank3 N-PDZ之间的相互作用模式。
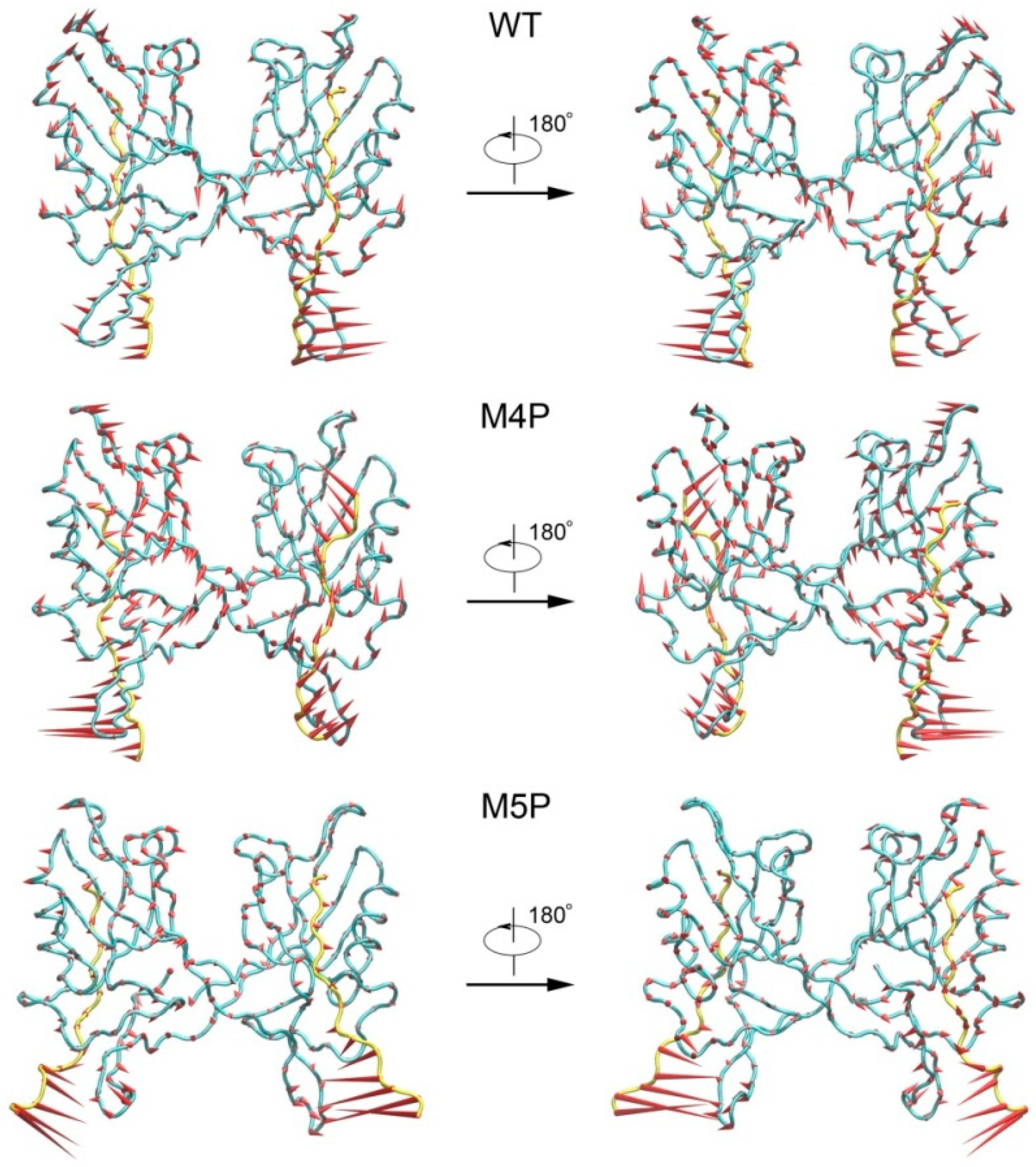
PCA分析用于提取系统的基本运动模式。结果显示,在野生型系统中,整体结构相对稳定,但βN发夹结构域表现出较大的运动幅度。在M4P和M5P系统中,突变导致E-PBM的相关部分从其原始结合位置偏离,进一步证实了突变对结合的影响。
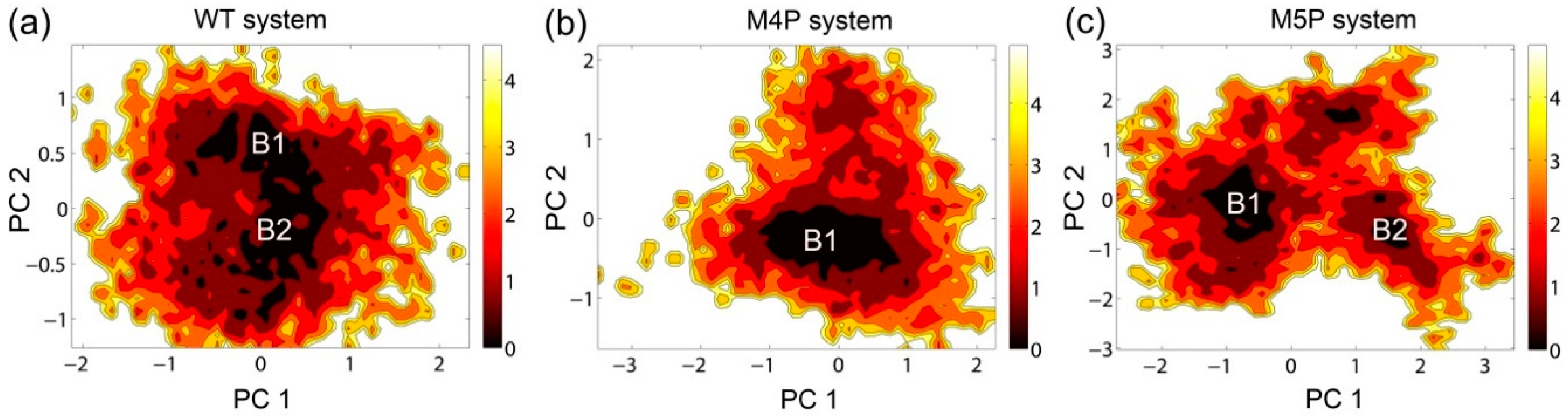
自由能景观(FEL)分析有助于揭示复合物在不同构象下的稳定性,颜色越深表示能量越低。通过FEL分析,能够识别出野生型和突变型SAPAP与Shank3复合物的稳定构象。研究发现,突变系统的PC1和PC2运动模式比WT系统的范围更大,这表明突变引起了构象重排。
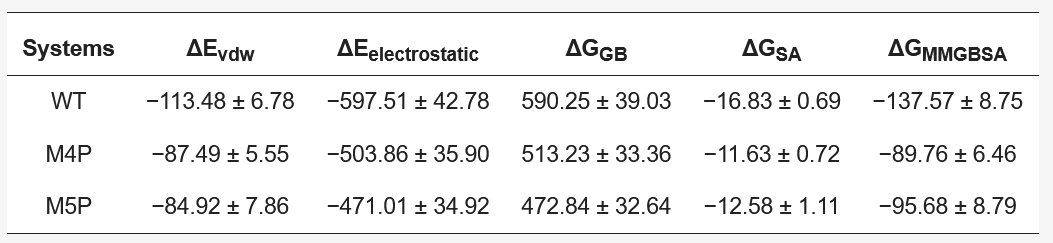
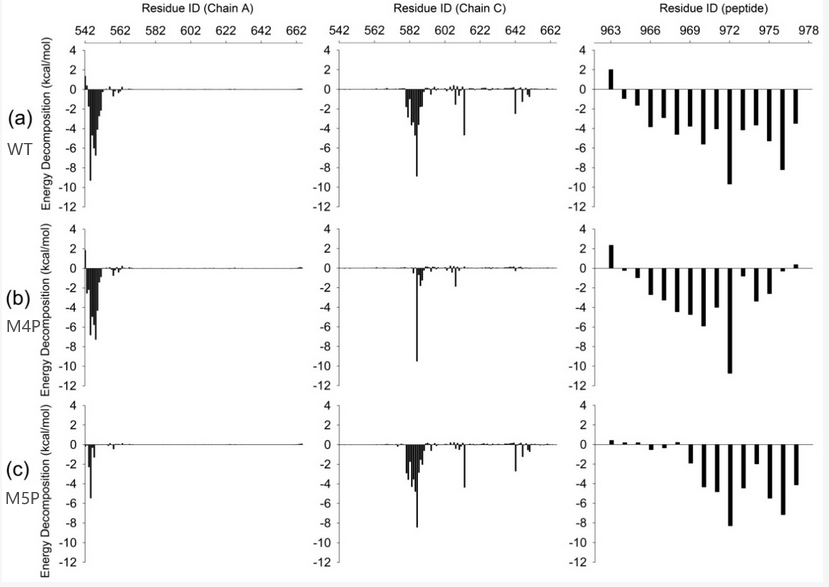
MM/GBSA方法用于计算结合自由能,以评估野生型和突变型E-PBM与Shank3 N-PDZ之间的结合亲和力。结果显示,野生型系统的结合自由能最低,表明其结合亲和力最高。相比之下,M4P和M5P系统的结合自由能增加,表明突变降低了结合亲和力。此外,研究人员还进行了能量分解,以评估每个残基对结合自由能的贡献。结果显示,突变残基的能量贡献显著降低,这与RMSF和PCA分析的结果一致。
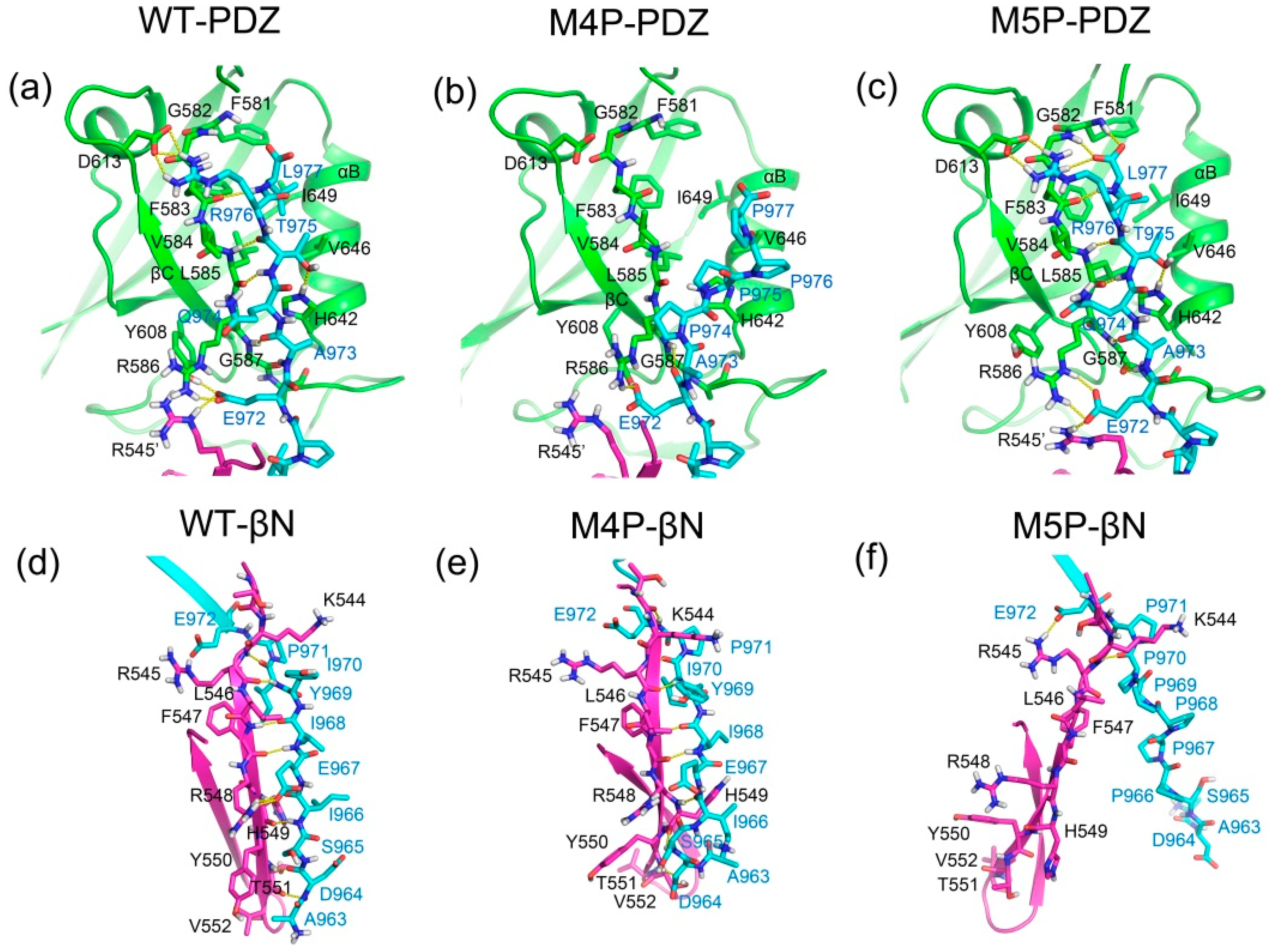
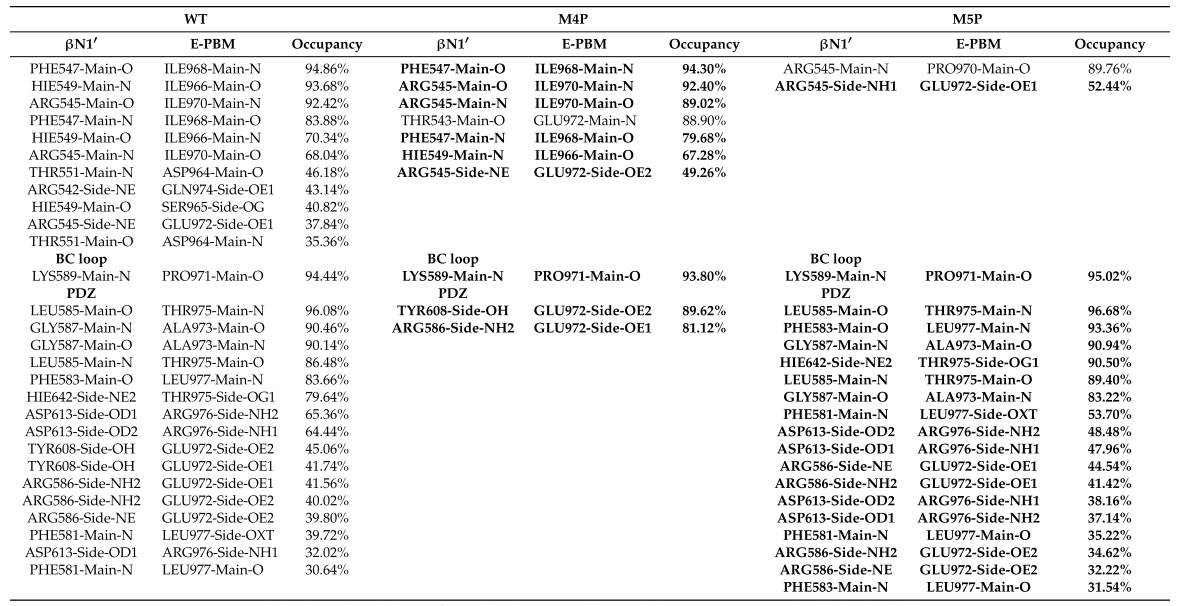
从结合模式和氢键占有率可以看出,相较于野生型,M4P突变导致canonical PDZ domain中大部分氢键的丢失,与βN1’的相互作用仍然存在。M5P突变消除了E-PBM与βN1’的相互作用,但与野生型相比,与PDZ结构域的相互作用保持良好。与能量计算结果一致,突变破坏了E-PBM与N-PDZ之间的相互作用,导致其结合能力降低。
本研究通过MD模拟和MM/GBSA结合自由能计算,深入研究了SAPAP野生型和突变型E-PBM与Shank3 N-PDZ之间的相互作用机制。结果表明,突变影响了E-PBM与Shank3 N-PDZ之间的相互作用模式,降低了结合亲和力。这些发现有助于理解SAPAP和Shank3特异性结合的分子机制,并为设计肽抑制剂干扰它们之间的相互作用提供了线索。该研究成果已发表于学术期刊《International journal of molecular sciences》。
Piao L, Chen Z, Li Q, et al. Molecular dynamics simulations of wild type and mutants of SAPAP in complexed with Shank3[J]. International journal of molecular sciences, 2019, 20(1): 224.